インシリコデザインプロジェクト
1. メンバー
| プロジェクトリーダー | 李 秀栄 |
|---|---|
| 特任研究員 | 陳 怡安、二島 渉 |
| 研修生 | ピロズマンド フィルゼ |
| 事務補助員 | 東條 充代 |
2. 研究目的・背景
本プロジェクトでは、コンピュータ解析により、疾患に関わる多数の生体分子やその反応を「機能を生み出す一つのシステム」として理解し、それに基づく医薬品のデザインを目指しています。インフォマティクスを活用して、創薬標的周辺のシステムバイオロジーの理解を通した標的の選択性を高めるとともに、医薬品候補の薬効・動態・毒性を予測し、効率的な選定・デザインを達成することを目指しています。
3. 研究内容
「分子認識」と「生体応答」という二つの重要な生物学的機能の解明に向けた研究に取り組んでいます。また、独自に開発した予測やデータ解析技術に基づいて実験研究者に仮説を提示し、実験による検証からのフィードバックを受けて新たな仮説の提示をするといった「仮説−検証サイクル」を確立しています。
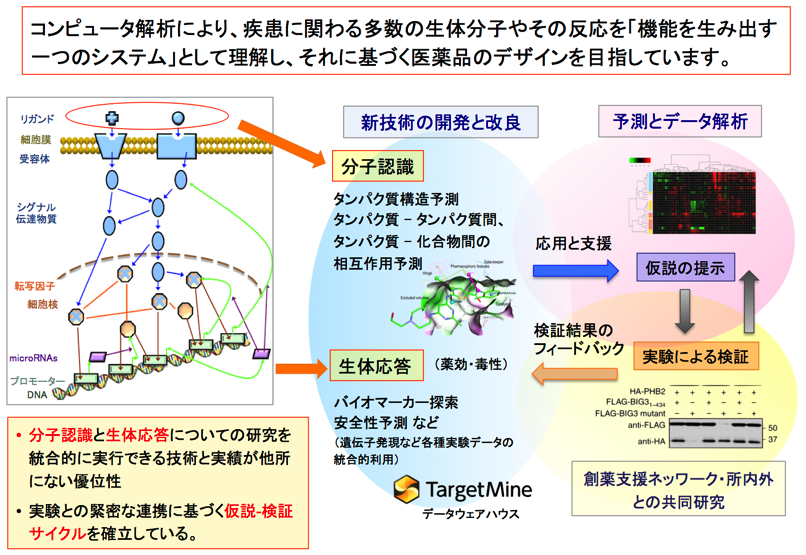
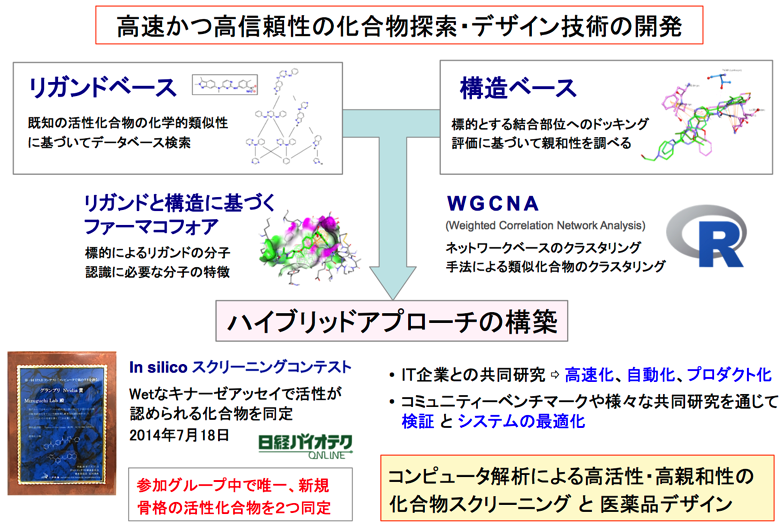
3-1. 分子認識に関する研究実例
リガンドベースと構造ベースのスクリーニング法を組み合わせた独自のスクリーニング法を確立しています。2014年に開催された「In silicoスクリーニングコンテスト」では、主催者側が用意した約240万化合物を収載した化合物ライブラリーの中から、参加グループの中で唯一、標的蛋白質(ヒトのc-Yes kinase)の活性を阻害する新規の骨格を持つ活性化合物を2つ同定することに成功しました。
(参考文献)
- Prathipati P, Mizuguchi K, Integration of Ligand and Structure Based Approaches for CSAR-2014, J. Chem. Inf. Model, 2015.
- Prathipati P, Mizuguchi K, Systems Biology Approaches to a Rational Drug Discovery Paradigm, Current Topics in Medicinal Chemistry, 16(9):1009-1025, 2015.
- Chiba S, Ikeda K, Ishida T, Gromiha M, Taguchi Y, Iwadate M, Umeyama H, Hsin K, Kitano H, Yamamoto K, Sugaya N, Kato K, Okuno T, Chikenji G, Mochizuki M, Yasuo N,Yoshino R, Yanagisawa K, Ban T T, Teramoto R, Ramakrishnan C, Thangakani A. M, Velmurugan D, Prathipati P, Ito J, Tsuchiya Y, Mizuguchi K, Honma T, Hirokawa T, Akiyama Y, Sekijima M, Identification of potential inhibitors based on compound proposal contest: Tyrosine-protein kinase Yes as a target, Scientific Reports, 5:17209, 2015.
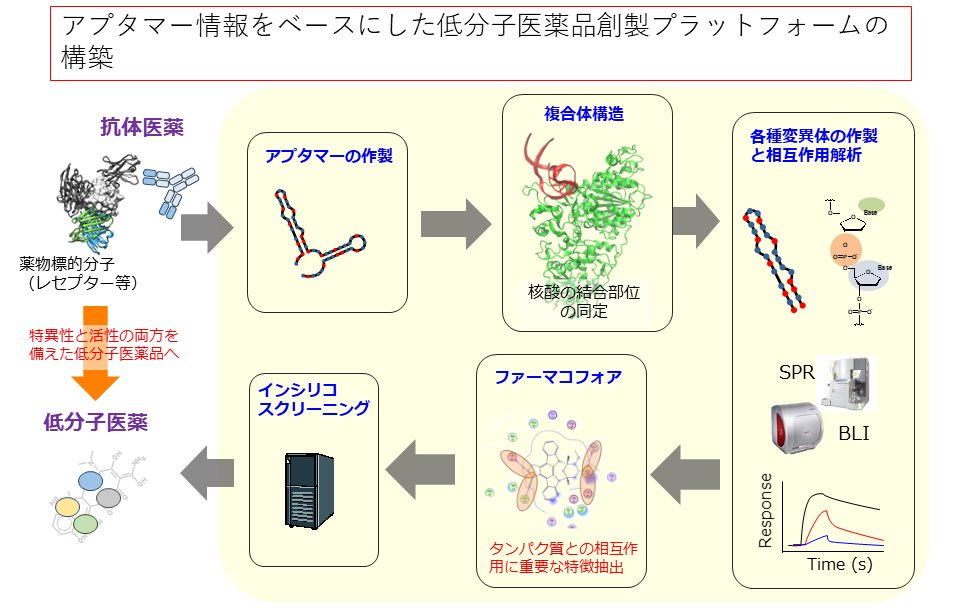
近年、抗体医薬品などの標的に対して高い特異性を持つバイオ医薬品が台頭し、様々な疾患に対し新たな治療法がもたらされた一方で、医療費の増大や経口投与が難しいため患者の生活の質の向上が必要といった課題が生じています。
そこで、これらの課題を解決すべく、バイオ医薬品の優れた生理機能を論理的に低分子化合物に変換する技術の開発を行っています。
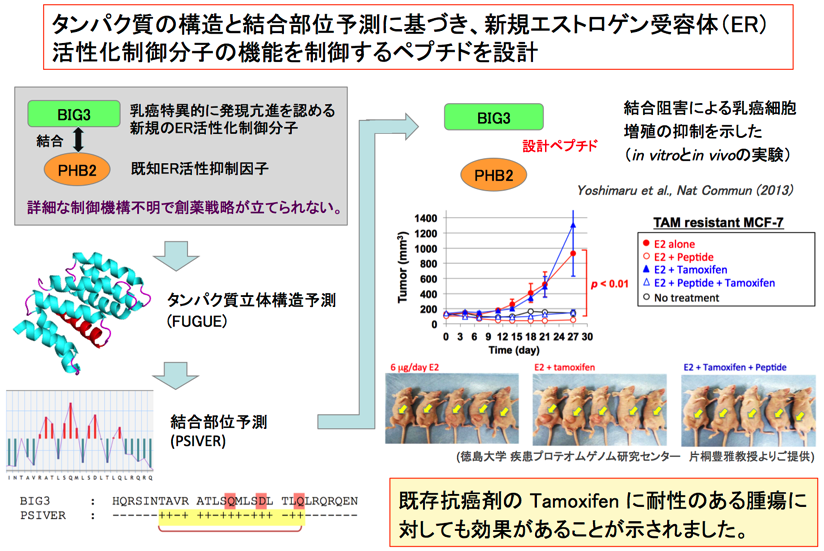
創薬候補蛋白質の構造や機能に関する情報がなく、実験的に先に進めることができない場合があります。そのような壁を越えるために、蛋白質の配列情報のみから立体構造や他の蛋白質と相互作用する残基を予測する手法を開発しました。これらの手法を新規のエストロゲン受容体活性制御分子BIG3に適用して、予測した部位のペプチドを合成し、マウスのモデルで既存抗癌剤に耐性にある腫瘍に対しても増殖抑制能を示すことが示されました。
(参考文献)
- Murakami Y, Mizuguchi K. Applying the Naive Bayes classifier with kernel density estimation to the prediction of protein-protein interaction sites. Bioinformatics, 26:1841-1848, 2010.
- Yoshimaru T, Komatsu M, Matsuo T, Chen YA, Murakami Y, Mizuguchi K, Mizohata E, Inoue T, Akiyama M, Yamaguchi R, Imoto S, Miyano S, Miyoshi Y, Sasa M, Nakamura Y, Katagiri T. Targeting BIG3-PHB2 interaction to overcome tamoxifen resistance in breast cancer cells. Nat Commun, 4:2443, 2013.
- Yoshimaru T, Ono M, Bando Y, Chen YA, Mizuguchi K, Shima H, Komatsu M, Imoto I, Izumi K, Honda J, Miyoshi Y, Sasa M, Katagiri T, A-kinase anchoring protein BIG3 coordinates oestrogen signalling in breast cancer cells, Targeting BIG3-PHB2 interaction to overcome tamoxifen resistance in breast cancer cells. Nat Commun, 8:15427, 2017.
3-2. 生体応答の理解に関する研究実例
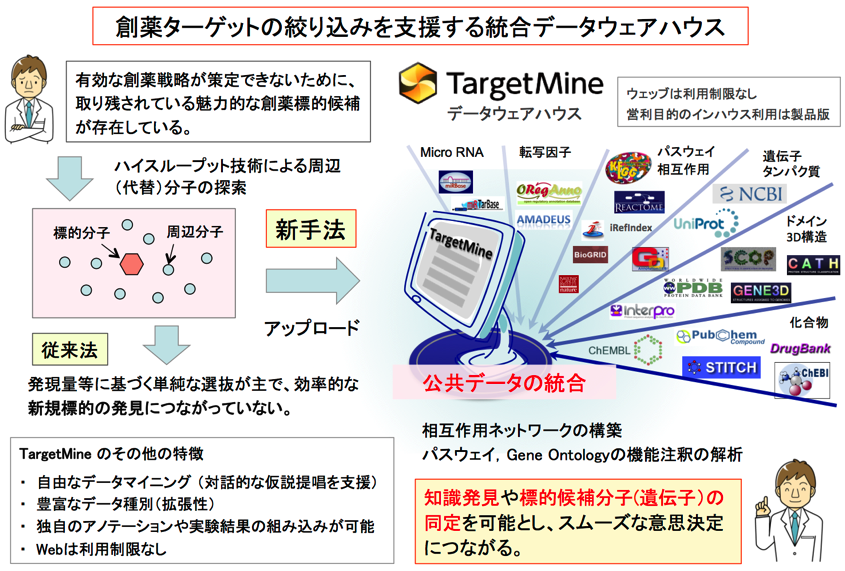
有効な創薬戦略が策定できないために取り残されている魅力的な創薬標的候補が存在します。そこで、公共的に利用可能な生命科学に関するデータを統合したTargetMineデータウェアハウスを開発しました。これは、対象とする疾患や生命現象に関わる標的候補周辺のネットワークの探索的な解析を行うことで、より有効な標的選定と実験的評価を可能とするシステムです。
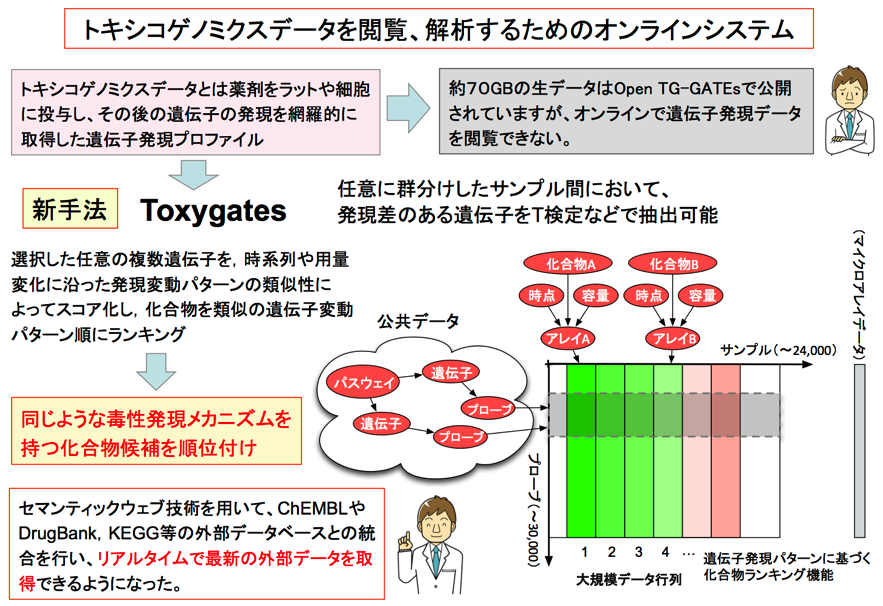
また、セマンティックウェブ技術を用いて、トキシコゲノミクスデータと外部データベースとの統合を行い、リアルタイムに解析可能な統合データ解析プラットフォームを開発、公開しています。
(参考文献)
- Chen YA, Tripathi L.P, Mizuguchi K, TargetMine, an integrated data warehouse for candidate gene prioritisation and target discovery, PLoS One, 8;6(3):e17844, 2011.
- Tripathi LP, Kambara H, Chen YA, Nishimura Y, Moriishi K, Okamoto T, Morita E, Abe T, Mori Y, Matsuura Y, Mizuguchi K. Understanding the biological context of NS5A-host interactions in HCV infection: a network-based approach. J Proteome Res, 12:2537-2551, 2013.
- Chen YA, Tripathi LP, Dessailly BH, Nystrom-Persson J, Ahmad S, Mizuguchi K. Integrated pathway clusters with coherent biological themes for target prioritisation. PLoS One, 9:e99030 , 2014.
- Chen YA, Tripathi LP, Mizuguchi K, An integrative data analysis platform for gene set analysis and knowledge discovery in a data warehouse framework, Database (Oxford), 2016.
(参考論文)
- Nyström-Persson J, Igarashi Y, Ito M, Morita M, Nakatsu N, Yamada H, Mizuguchi K. Toxygates: interactive toxicity analysis on a hybrid microarray and linked data platform. Bioinformatics, 29:3080-3086, 2013.
- Nyström-Persson J, Natsume-Kitatani Y, Igarashi Y, Satoh D, Mizuguchi K, Interactive Toxicogenomics: Gene set discovery, clustering and analysis in Toxygates. Sci Rep., 7(1):1390, 2017.
- Natsume-Kitatani Y., Nystrom-Persson J., Igarashi Y., Satoh D., Mizuguchi K., Integrated toxicogenomics analysis with Toxygates for inferring molecular mechanisms. GENOMICS AND COMPUTATIONAL BIOLOGY, 3(1):e37, 2017.
3-3. 創薬デザインのための基盤ツールの開発例
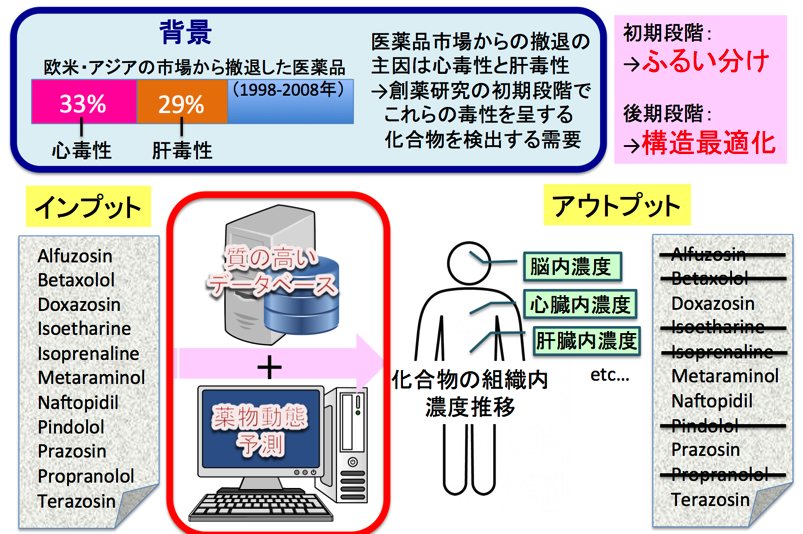
効率的な医薬品候補創出のためには、薬物の吸収・分布・代謝・排泄といった体内動態及び毒性の予測を目的としたインシリコの統合解析プラットフォームを構築することが必要であると考えています。対象化合物の化学構造から各種物性パラメータ(薬物の膜透過性や血漿蛋白質結合性など)を予測する構造活性相関モデルを開発し、さらにこれらの予測結果を基に薬物動態を高性能で予測できるシステムの構築を目指した研究を進めています。
(その他の主要論文)
- Esaki T, Watanabe R, Kawashima H, Natsume-Kitatani Y, Mizuguchi K, Development of a Drug Discovery Informatics System: An Integrated Platform for Pharmacokinetic and Toxicological Modelling. J. Pharm. Sci. Technol.77(4): 211-215, 2017.
